欧洲分子生物学实验室研究人员Moritz Kueblbeck, Andrea Callegari等在bioRxiv分享了一项基于CRISPR方法优化的技术文章,目的在于提升从基因编辑产生的细胞中筛选纯合子的效率。众所周知,使用CRISPR精确敲入 (knock-in) 外源性DNA后,产生的基因编辑克隆需要严格的筛选才能得到目的克隆(纯合子)。基于常规PCR和Southern Blot检测目的克隆的方法耗时长、需要大量劳动力。因此研究人员对传统方法进行了优化,采用荧光标记蛋白和naica数字PCR联合的方法,精准快速的实现了基因编辑后目的克隆的筛选,大大降低了检测的人力、物力、时间成本。
亮点:
☑ 采用数字PCR和荧光标记的方法,可以在基因组编辑的早期阶段进行检测,整个流程只需要大约3-4小时即可获得结果,能够及时停止“不理想”的编辑克隆的培养,节省人力物力成本。
☑ 数字PCR方法只需要检测少量的细胞,相较于Southern Blot大大减少样本制备时间。
☑ 基于naica®微滴芯片数字PCR系统的多重检测能力和Crystal Miner软件的荧光补偿校正功能,能够快速、可靠的对CRISPR编辑细胞进行定量筛选检测。
文章内容分享
背景原理介绍:
CRISPR(Clustered Regularly Interspaced Short Palindromic Repeats),规律间隔成簇短回文重复序列,是一种RNA引导的基因组编辑技术,能对基因进行精确性敲除,敲入,替换等,从而实现探究基因功能,修复致病基因等目的。但是,CRISPR技术也会导致有潜在危险的“脱靶”问题,带来不可预测的基因变化,因此对于CRISPR基因编辑的脱靶情况需要灵敏且精准的验证及检测。
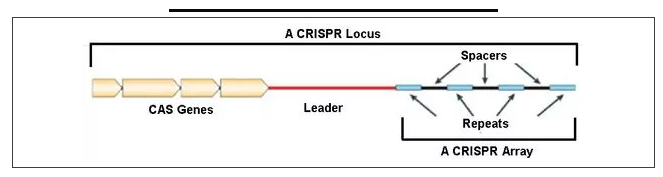
▲CRISPR结构示意图
研究目的:
提升从基因编辑产生的细胞中筛选纯合子的效率。
传统方法筛选目的克隆的局限性:
基因编辑后,筛选纯合子的方法是生成杂合克隆后,通过轮次迭代产生纯合子,整个过程耗时甚至能长达一年,且容易发生脱靶修饰。使用常规PCR区分纯合克隆和杂合克隆的方法只能检测到目的基因,还需要金标准Southern Blot检测脱靶整合的情况,然而,Southern Blot是一种耗时且低通量的技术,需要相对大量的基因组DNA和细胞材料,这些材料的制备会浪费大量的时间和人力成本。
优化后的新方法:
针对上述问题,Moritz Kueblbeck等人优化了CRISPR的实验流程,改进了CRISPR的转染效率,通过添加荧光标签蛋白实现CRISPR编辑的克隆菌落中相关标记蛋白的正确亚细胞定位,并通过dPCR 来定量目的基因和脱靶基因组编辑事件。通过该方法,快速、可靠的对荧光标记CRISPR编辑细胞进行了定量筛选。【见下图】
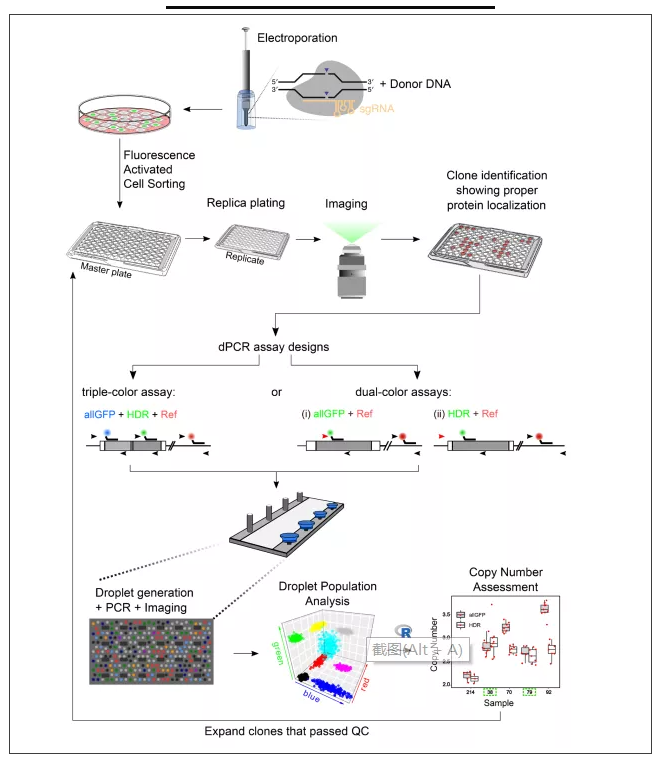
▲Moritz Kueblbeck等人优化的CRISPR纯合子筛选实验流程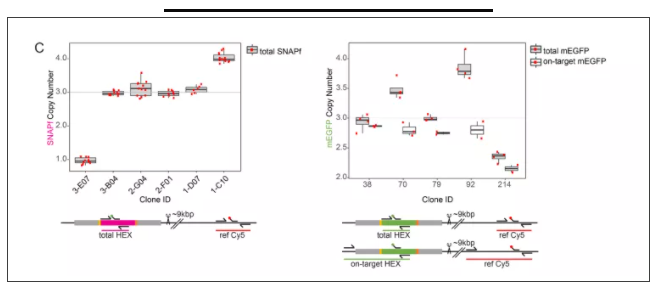
▲PCR进行两种细胞系中的标签基因检测。U2OS- TPR-SNAP标签基因整合总数检测 (左);
HK- Nup93-mEGFP标签基因整合总数及目的标签基因检测(右)。矩形图代表克隆,每个红点代表一次测量结果。
对于多重荧光检测实验,进行荧光溢出的补偿校正是十分必要的。本研究使用naica®微滴芯片数字PCR系统进行多重检测,并使用Crystal Miner软件进行荧光补偿校正分析,确保每一个荧光基团被单一检测通道捕获,得到的结果精准可靠。
如想对荧光补偿校正有更多了解,请复制下方链接地址到浏览器进行查看:http://dx.doi.org/10.1016/j.bdq.2016.10.002

原文链接如下:https://doi.org/10.1101/2021.06.23.449557
CRISPR WEBIANR相关视频链接:
▲点击上方图片观看相关视频
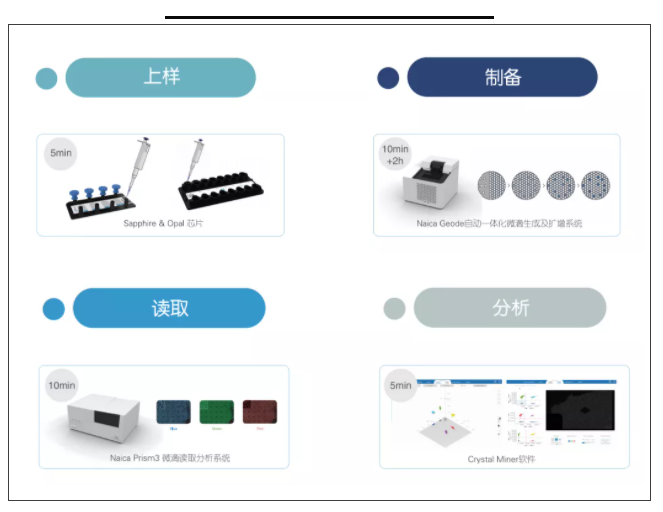
naica®微滴芯片数字PCR系统
法国Stilla Technologies公司的naica®微滴芯片数字PCR技术在进行核酸检测时具有独特的优势。系统利用cutting-edge微流体创新型芯片—Sapphire芯片(或高通量Opal芯片)作为数字PCR过程的唯一耗材。样品通过毛细通道网格以30,000个微滴的形式进入2D芯片中。3色荧光检测仪器,整个流程只需要两个半小时,并可进行数据的质控和结果追溯分析,获得的数据真实可靠。