

废水中检测到新冠病毒和新冠变种,在世界各地的污水厂都有类似的报道。今年初,美国疾病控制预防中心 (CDC)发布的报告中,指出新冠病毒变种奥密克戎(Omicron)很有可能在美国首例确诊病例官方声明前一周已经存在于纽约市的废水中,报告同时指出,废水监测是一个可行的早期预警系统,可以帮助跟踪新冠病毒变种的传播。
尽管对废水中的SARS-CoV-2 RNA测序的方法极具应用意义,可以从单个样本中追踪数百人到数千人的新冠变种VOCs,但在样本收集和分析方面存在时间滞后,测序和生物信息学管道通常需要至少3天的时间才能得到结果,为了能更快速地监测,科学家们开发了基于PCR的分析方法,以识别其特征突变(Vogels等人,2021;Bedotto等人,2021;Chaintoutis等人,2021)。例如,针对单核苷酸多态性的检测,如突刺蛋白中的N501Y和E484K,或特征缺失,如突刺Δ69-70和ORF1a Δ3675- 3677。
瑞士科学家Lea Caduff等使用drop-off数字PCR方法开发快速、高通量的方法来检测和量化废水中新冠变种的两种缺失频率,开发统计方法监测特异性突变随时间的动态变化,从而量化新冠变种的传播速度,数据与临床样本测序结果一致。该方法基于naica®微滴芯片数字PCR系统,针对废水中新冠病毒特异性突变的数字PCR检测提供了近实时的SARS-CoV-2 新冠变种VOCs监测,并可能比临床测序更早地检测和推断新冠变种VOCs的传播速度,方法发表于文章“Inferring Transmission Fitness Advantage of SARS-CoV-2 Variants of Concern in Wastewater Using Digital PCR。DOI:https://doi.org/10.1101/2021.08.22.21262024。
应用亮点:
▶ 利用naica®微滴芯片数字系统,开发出drop-off方法检测废水中SARS-CoV-2变种的突变频率。
▶ 通过对大量废水及临床样本的检测分析,发现废水中SARS-CoV-2变种的传播适应性与临床样品一致。
▶ Drop-off RT-dPCR方法为社区内VOCs的快速检测和定量提供了机会,可用于基于SARS-CoV-2的废水流行病学的大规模检测调查。
▶ 与临床或废水测序相比,drop-off RT-dPCR检测提供了一种在社区内跟踪突变的方法,成本更低,检测速度更快,而且可以用于筛选VOCs的其它突变。
在COVID-19全球大流行期间,SARS-CoV-2令人担忧的遗传变异(VOCs)多次出现。VOCs的特征是传播性增强,毒性增强,或通过先前感染或接种疫苗获得的抗体的中和作用减弱。通常追踪VOCs的输入和传播依赖于测序,对临床样本进行全基因组测序。现在废水监测越来越多地被用于通过测序方法跟踪SARS-CoV-2变种的输入和传播。虽然废水中SARS-CoV-2 RNA测序是一种很有前途的方法,可以在一个样本中跟踪成百上千个人的VOCs,但样本收集和分析存在时间滞后,这与临床测试相关的滞后类似。在得到RNA样本后,测序和生物信息学分析通常需要至少3天的时间才能得到结果。为了更快速地识别VOCs,我们开发了基于PCR的快速、高通量的方法来检测和定量废水中B.1.1.7、B.1.351和P.1 VOCs的两种突变频率。我们进一步开发了一种统计方法来分析RT-dPCR检测数据的时间动态,以量化传播适应性,获得与从临床样本获得的相似的数据。针对废水中的SARS-CoV-2数字PCR检测提供了对VOCs的实时监测,并可以比临床测序更早地发现和推断传播适应性。
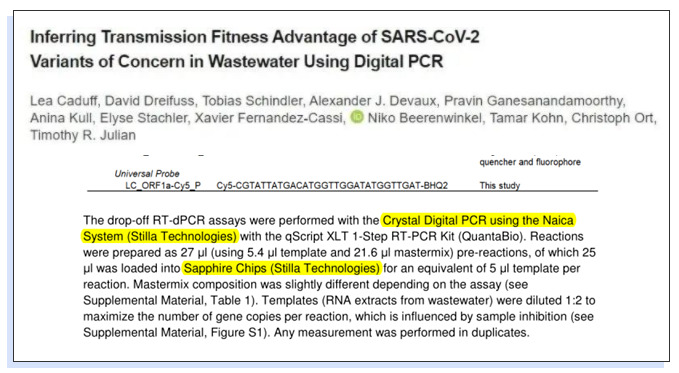
在一个RT-dPCR检测中,使用两个非竞争性水解探针,针对同一扩增子上的保守区域,量化废水样品中的突变频率(“drop-off RT-dPCR检测”),可以同时定量野生型(不携带突变的菌株)和突变型(携带突变的菌株)(图1)。
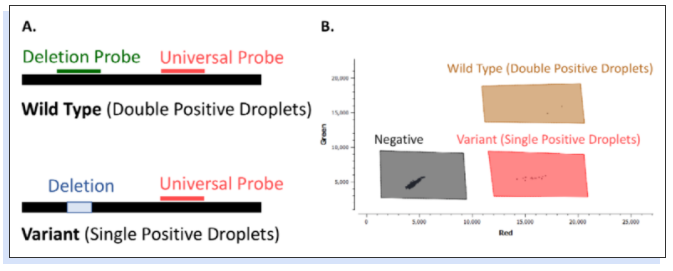
▲图1 基于两种不同探针的drop-off RT-dPCR检测方法概述:一种drop-off探针(Deletion Probe)和一种Reference探针(Universal Probe)。(A)Reference探针可同时与野生型和突变型结合,而drop-off探针只与野生型结合。(B)在dPCR 2D散点图中,野生型为双阳性液滴(棕色),而突变型为单阳性液滴(红色)。
在2020年12月7日至2021年3月26日期间,对从废水处理厂Werdhölzli(服务于瑞士苏黎世约45万人)收集的32个原始进水(24小时流量比例复合)样本进行了drop-off RT-dPCR检测,并与在苏黎世收集并测序的2497个临床样本进行对比。使用drop-off RT-dPCR方法在废水中检测的突变的时间趋势与临床样本的测序数据一致(图2)。
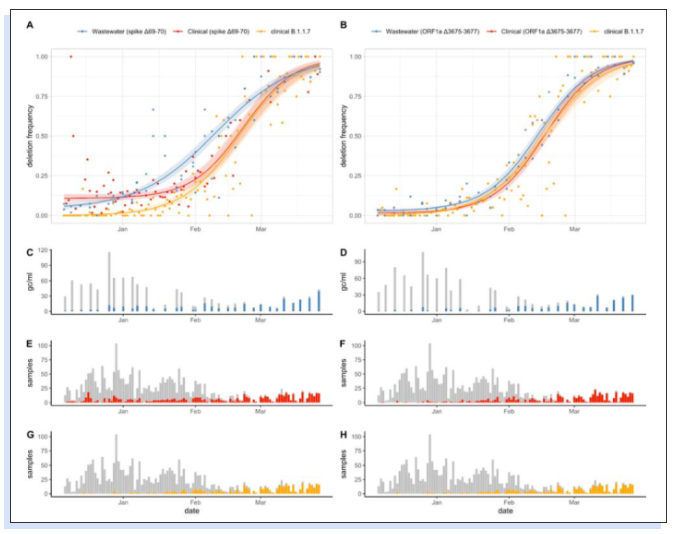
▲图2 与苏黎世临床样本相比,废水样本中 (A) spike Δ69-70 和 (B) ORF1a Δ3675-3677 的突变频率,以及苏黎世临床样本中 B.1.1.7 谱系的突变频率。拟合曲线对应于废水和临床数据缺失的三参数拟合和 B.1.1.7 临床数据的两参数拟合。阴影区域对应于 95% 的置信带。(C) spike Δ69-70 和 (D) ORF1a Δ3675-3677 的 SARS-CoV-2 RNA(灰色)浓度和缺失等位基因(蓝色)浓度,在废水样品中。(E) spike Δ69-70 和 (F) ORF1a Δ3675-3677,苏黎世测序的临床样本数量(灰色)和缺失等位基因(红色)的样本数量。(G, H) 苏黎世测序的临床样本数量(灰色)和B.1.1.7 谱系的样本数量(橙色)。
使用dPCR技术跟踪废水中VOCs的特征突变(ORF1aΔ3675-3677和spikeΔ69-70)的时间趋势可以迅速告知变异传播性。来自废水的生长速率和传播适应性估计值与来自临床样本的估计值基本一致,95%置信区间重叠。由于dPCR样本分析比全基因组测序更快,一旦识别出特征突变,可以帮助我们早期洞察新变异的传播性。本研究可以帮助大家进行SARS-CoV-2变种的传播性研究。
参考文献:
1、Bedotto, Marielle, et al. 2021. “Implementation of an in-House Real-Time Reverse Transcription-PCR Assay for the Rapid Detection of the SARS-CoV-2 Marseille-4 Variant.” Journal of Clinical Virology: The Official Publication of the Pan American Society for Clinical Virology 139 (June): 104814.
2、Chaintoutis, et al. 2021. “A One-Step Real-Time RT-PCR Assay for Simultaneous Typing of SARS-CoV-2 Mutations Associated with the E484K and N501Y Spike Protein Amino-Acid Substitutions.” medRxiv. https://doi.org/10.1101/2021.05.31.21257367.
3、Fernandez-Cassi, et al.. “Wastewater Monitoring Outperforms Case Numbers as a Tool to Track COVID-19 Incidence Dynamics When Test Positivity Rates Are High.” https://doi.org/10.1101/2021.03.25.21254344.
4、Crits-Christoph, et al. 2021. “Genome Sequencing of Sewage Detects Regionally Prevalent SARS-CoV-2 Variants.” mBio 12 (1). https://doi.org/10.1128/mBio.02703-20.
