

导读
汉堡-埃彭多夫大学医学中心(UKE)的研究者在Molecular Therapy: Methods & Clinical Development上发表了题为LATE–a novel sensitive cell-based assay for the study of CRISPR/Cas9-related long-term adverse treatment effects的文章。
文中应用流式细胞术和naica®微滴芯片数字PCR系统开发了一种长期不良治疗效应(LATE)体外检测方法评估促生长脱靶事件的风险,通过细胞转化事件来评估Cas9蛋白的潜在毒性。在小规模原理验证实验中,可重复地检测到由TP53基因脱靶切割引起的原代人新生儿包皮成纤维细胞(NUFF细胞)中低频的(<0.5%)促生长事件。作者还比较了不同Cas9蛋白的脱靶毒性,证明现有的高保真核酸酶比第一代Cas9脱靶概率大幅降低。在这项概念验证研究中,LATE检测可以评估CRISPR/Cas9系统的生理学不良反应,有利于临床前安全性研究。

自CRISPR / Cas9技术问世以来,基因编辑已广泛应用于基础研究和应用生物学领域,将其转化为临床试验则引起了安全性问题。尽管脱靶事件的频率和位置已被广泛研究,但人们对它们在大规模、长期的应用环境中潜在的生物学后果知之甚少。所以开发一种操作性强的体外检测方法来评估这一问题则至关重要。
由于脱靶相关的风险非常明显,这不仅促成了各种算法的建立来预测靶位点脱靶突变的可能性,而且还建立了几种无偏、通用的技术来分析全基因组的双键断裂(DSB)。但现有的主流方法都依赖于某种形式的高通量测序来检测靶位点或全基因组范围内的突变。而二代测序(NGS)是一种相当昂贵的方法,也需要先进的生物信息学技术来进行详细的数据分析。此外,上述所有算法和方法仅能提供脱靶的位置和频率信息,而它们对细胞生理学的潜在后果仍是未知。目前也缺乏对设计核酸酶Cas9蛋白潜在长期不良影响的评估方法。
应用亮点:
▶ 使用naica®微滴芯片数字PCR系统构建了GEF-dPCR(基因编辑频率数字PCR)检测方法。
▶ dPCR在检测低频突变时比NGS具有更高灵敏度和准确性。
▶ GEF-dPCR可以和NGS互相验证,GEF-dPCR可能比NGS更不容易受到单核苷酸缺失或交换的影响。
实验方法:
之前有研究表明NUFF细胞敲除TP53会导致相对生长优势,作者根据这种已经确立的生长优势设计了图1所示的LATE检测流程。LATE检测的步骤如下:
1、通过编码eGFP、 Cas9和gRNA的一体化慢病毒载体LeGO-CC转导NUFF细胞;
2、转导8-10周后每周通过流式细胞术分析细胞的荧光强度;
3、通过定量转导细胞生长优势指标来判断基因组编辑的影响,阳性结果中荧光细胞占绝对优势,阴性结果中荧光细胞不存在生长优势。
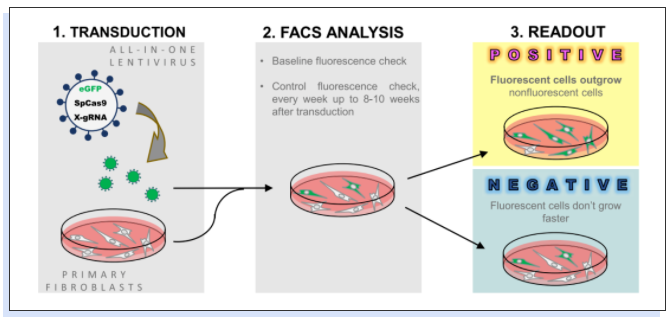
▲图1. LATE检测原理图
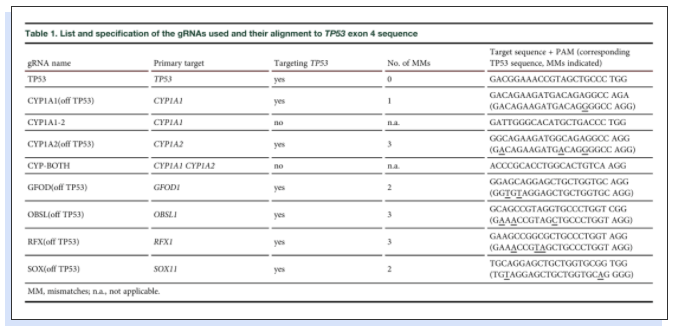
为了确定LATE检测是否也可用于评估由非预期脱靶TP53敲除引起的生长优势,作者筛选了5个靶向其他基因但与TP53具有一定相似性的gRNA,它们含有1到3个与TP53编码序列的错配。如表1所示,CYP1A1/2、GFOD、OBSL和RFX gRNA会有一定概率靶向TP53,引起NUFF细胞的生长优势。
表1.研究使用的gRNA列表及其与TP53外显子4序列的比对
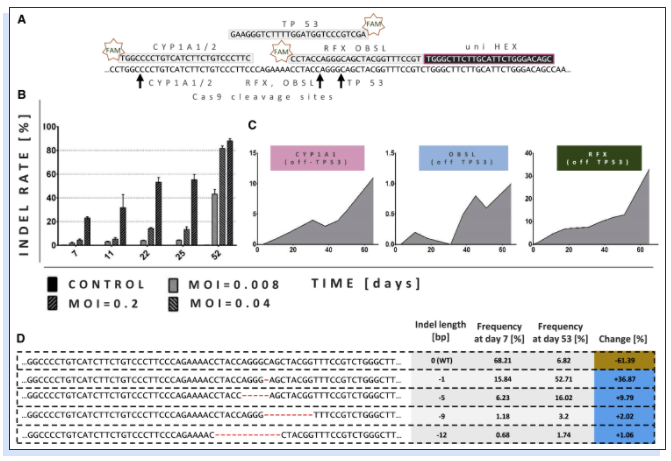
LATE检测中的阳性结果理论上也可能是由非预期脱靶TP53敲除以外的原因(如插入突变)引起的。为了研究LATE检测的阳性结果和(从头产生的)TP53插入/缺失(indel)之间的联系,作者使用naica®微滴芯片数字PCR系统设计了一种新型的GEF-dPCR技术,又被称为drop-off检测。该方法使用两个双标记的水解探针,可用于定量Cas9介导的DSB修复期间由非同源末端连接(NHEJ)引起的gRNA结合位点的indel频率。HEX标记的探针结合区域远离gRNA识别序列,因此其结合不受DNA修复过程影响。FAM标记drop-off探针的结合会被NHEJ产生的indel影响。

实验结果:
作者应用GEF-dPCR来量化编码Cas9和CYP1A1、OBSL和RFX gRNA的LeGO CC载体转导NUFF细胞中脱靶至TP53引入的indel频率。他们使用了相同的PCR引物和HEX标记的探针,并设计了FAM标记的靶标特异性drop-off探针(图3A)。结果显示三种gRNA的阳性LATE检测结果确实与TP53 indel的增加相关(图3C)。值得注意的是,在转导OBSL gRNA的情况下,可检测到的TP53 indel频率低至0.2%(图3C),这种低频的脱靶效应可以通过LATE检测来定量,并且在所有对照样本中没有检测到类似的TP53外显子4 突变和indel。这一观察凸显了LATE检测在发现罕见的促生长事件方面的敏感性,比NGS具有更高灵敏度和准确性。
点击图片查看大图
▲图3 NUFF细胞获得的生长优势与TP53中的indel频率相关。(A)用于GEF-dPCR的TP53外显子4和互补的FAM和HEX标记探针片段如图所示。FAM标记的探针结合gRNA靶向Cas9的位置,因此当TP53序列的互补区域内没有indel时能够结合。HEX标记探针结合Cas9切割位点外的TP53序列。(B)在指定时间点从NUFF细胞中获得基因组DNA样品,这些细胞在三个不同的MOI下转导了编码eGFP,Cas9和靶向TP53 gRNA(LeGO-CC-p53)的一体化慢病毒颗粒(LeGO-CC-p53)。通过GEF-dPCR定量一段时间内靶向TP53外显子4的indel频率。(C)在指定的时间点从NUFF细胞中获得基因组DNA样品,这些细胞转导了编码靶向Cas9及CYP1A1,OBSL1或RFX1的gRNA一体化慢病毒颗粒。通过GEF-dPCR定量一段时间内非预期脱靶TP53外显子4敲除的indel频率。(D)用LeGO-CC-p53治疗后第7天和第53天对TP53外显子4进行深度测序得到的indel长度及频率。
为了增加CRISPR/Cas9系统的靶向特异性,Cas9核酸酶一直是优化的重点。科学家对SpCas9一直在进行结构设计的修改以及随机诱变。有研究表明正电荷残基(K855A)的单个丙氨酸取代可以降低Cas9脱靶活性。作者通过病毒转导靶向TP53分析SpCas9 K855A的核酸酶特异性。如图6A所示,在实验前10天,经SpCas9 K855A和wt Cas9处理细胞的eGFP阳性细胞生长动力学接近,但随着时间的推移SpCas9 K855A处理的eGFP阳性细胞数量更多。作者通过GEF-dPCR和深度扩增子测序这两种技术都证实,随着时间的推移TP53中携带indel的细胞数量不断增加。GEF-dPCR检测到转导52天后最高插入indel频率为65%(图6B),在转导后第55天约80%的NGS读数检测到indel。有趣的是当SpCas9 K855A与CYP1A1(非靶向TP53)gRNA一起使用时,与用相同gRNA和wt Cas9处理的NUFF细胞相比,eGFP阳性细胞的比例生长慢得多(图6A),这应该与SpCas 9 K855A特异性的提高有关。
在第二组实验中,作者对eSpCas9 1.0(特异性优于SpCas 9 K855A)进行了上述相同的分析,在LATE试验中没有观察到CYP1A1 gRNA脱靶TP53的eGFP阳性细胞的生长,而靶向TP53的gRNA与wt Cas9一样有效(图6A)。

点击图片查看大图
▲图6 LATE检测区分具有不同保真度的 SpCas9变体。(A)NUFF细胞与编码野生型wt SpCas9(灰色)或存在突变位点的SpCas9(黑色)的多合一LeGO-CC颗粒转导的NUFF细胞的FC分析比较。红线标记编码突变 SpCas9 变体的载体的初始转导率。阳性和阴性分别用黄色和蓝灰色表示。(B) 通过 GEF-dPCR 检测靶向TP53和非靶向 TP53(CYP1A1) 外显子 4的indel随时间推移的相对数量。
结论:
这里介绍的LATE检测结合GEF-dPCR技术可以填补基因编辑中的部分空白,作为一种简单,快速和高性价比的方法,用于测试给定基因组编辑策略对细胞生长调节的不良影响,评估不同的Cas9核酸酶在基因组编辑时的特异性。
原文链接如下:https://www.ncbi.nlm.nih.gov/pmc/articles/PMC8399046/#bib33
期刊介绍:
《molecular therapy》是发布分子和细胞疗法在纠正遗传和获得性疾病研究成果的国际领先期刊。文章涉及基因转移和编辑、载体开发和设计、干细胞操作、疫苗开发、临床前靶点验证、安全性/有效性研究和临床试验方面等研究领域。
